Forschungsbericht 2011
Struktur und Funktion von Spleißosomen
Die Bildung von Proteinen in Zellen höherer Lebewesen ist ein komplexer, mehrstufiger Vorgang. Zunächst wird die genetische Information für ein Protein von der DNA in eine Arbeitskopie, die Präkursor-Boten-RNA (prä-mRNA), umgeschrieben. Diese RNA-Kopien können jedoch erst dann für die Proteinherstellung verwendet werden, wenn durch präzisen Zuschnitt die nicht benötigten internen Bereiche, sogenannte „Introns“, entfernt und die informationsrelevanten Bereiche, „Exons“, neu verknüpft worden sind. Dieser Reifungsprozess wird als mRNA-Spleißen bezeichnet. Erst diese reife mRNA kann aus dem Zellkern ins Zytoplasma transportiert werden und den Ribosomen als Matrize zur Bildung von Proteinen dienen. Diese Exon-Intron-Anordnung bietet dem Organismus große Vorteile: Durch alternatives Spleißen, das heißt durch wahlweises Verknüpfen unterschiedlicher Exons, können von einem Gen mRNAs für verschiedene Proteine hergestellt werden. Alternatives Spleißen stellt damit eine zusätzliche Ebene der Regulation der Genexpression dar, die die genetische Kapazität höherer Eukaryonten enorm erhöht. Dies erklärt auch, warum der Mensch mit nur etwas mehr als 20.000 proteinkodierenden Genen in seinem Genom auskommt.
Die mRNA-Spleißreaktion erfolgt in zwei Schritten über Phosphoester-Transferreaktionen und wird im Zellkern durch die molekulare Maschine des Spleißosoms bewerkstelligt. Spleißosomen setzen sich aus weit über 100 Proteinen und fünf kleinen RNA-Molekülen (den snRNAs U1, U2, U4, U5 und U6) zusammen und sind damit gigantische, sehr proteinreiche Nanomaschinen. Viele dieser Bausteine sind in stabilen Subkomplexen organisiert. So lagern sich z. B. ca. 50 spleißosomale Proteine mit den snRNAs zu RNA-Proteinpartikeln, den snRNPs (small nuclear ribonucleoproteins) U1, U2 und dem U4/U6.U5-tri-snRNP, zusammen.

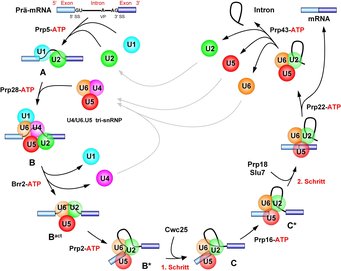
Spleißosomen existieren im Zellkern nicht als vorgefertigte Maschinen, sondern werden auf jedem zu spleißenden Intron jeweils von neuem aus ihren Bausteinen aufgebaut (Abb 1). Zunächst erkennen und binden die snRNPs U1 und U2 zusammen mit einigen Helferproteinen Anfang und Ende eines Introns. Die Bindung des U4/U6.U5-tri-snRNP führt dann zur Ausbildung des sogenannten B-Komplexes. Dieser Multi-Mega-Dalton-Komplex besitzt jedoch noch kein aktives katalytisches Zentrum. Schrittweise dramatische Strukturänderungen des B-Komplex-Spleißosoms verändern die Konformationen der snRNAs und die ursprüngliche biochemische Zusammensetzung des B-Komplexes. Dabei bildet sich ein komplexes Netzwerk von RNA-RNA-Wechselwirkungen zwischen der prä-mRNA und den snRNAs U2, U5 und U6 aus, das als Herzstück des katalytischen Zentrums zu betrachten ist (Abb. 2). Das katalytisch aktivierte Spleißosom ist jetzt in der Lage, den ersten Schritt der Spleißreaktion zu vollziehen, wobei das C-Komplex-Spleißosom entsteht. Nachdem der zweite katalytische Schritt erfolgt ist, wird das Spleißosom zusammen mit dem herausgeschnittenen Intron von der reifen mRNA abgelöst und in seine Einzelteile dissoziiert. Sowohl snRNAs als auch spleißosomale Proteine sind für die Funktion des Spleißosoms essenziell. So sind sie unter anderem an der Erkennung der Spleißstellen und der Ausbildung des katalytischen Zentrums beteiligt. Weiterhin sind eine Reihe energieverbrauchender Enzyme, sogenannte RNA-Helikasen, entscheidend an den schrittweise erfolgenden Umlagerungen des Spleißosoms beteiligt (Abb. 1).
Ein wesentliches Ziel der Forscher um Reinhard Lührmann liegt darin, die Funktionsweise und Struktur der Spleißmaschinerie zu verstehen. Dabei steht zum einen die Frage im Vordergrund, wie die Strukturumwandlungen des Spleißosoms während seines Arbeitszyklus vonstatten gehen und wie sie reguliert werden. Zum anderen wollen die Wissenschaftler die Frage klären, wie das katalytische Zentrum des Spleißosoms aufgebaut ist, d. h. besteht es wie bei einem Ribozym nur aus RNA-Bestandteilen oder agiert es eher wie ein RNP-Enzym, in dem RNA und Proteine gleichermaßen zur Katalyse beitragen? Um diese Fragen beantworten zu können, verwenden sie einen integrierten experimentellen Ansatz, der ein breites methodisches Spektrum umfasst. So analysieren sie die Funktion der Proteine und snRNA-Moleküle beim Spleißen mit biochemischen und molekulargenetischen Methoden, wobei die Forscher sich vornehmlich auf die Untersuchung von Spleißosomen aus Humanzellen und der Bäckerhefe konzentrieren. Gleichzeitig untersuchen sie mit der Kryo-Elektronenmikroskopie und der Röntgenkristallografie sowie mit massenspektrometrischen und fluoreszenzspektroskopischen Methoden die Raumstruktur und strukturelle Dynamik isolierter Spleißosomen.
Spleißosomen sind proteinreiche RNP-Maschinen mit sehr dynamischer biochemischer Zusammensetzung
Die biochemische Zusammensetzung der Spleißmaschinen ändert sich während eines Arbeitszyklus, d. h. während des Spleißens eines einzigen Introns, kontinuierlich. Aus Zellen kann man also keine endogenen biochemisch einheitlichen Spleißosomen isolieren. Es mussten daher Methoden entwickelt werden, die es erlauben, biochemisch einheitliche Funktionszustände eines Spleißosoms zu isolieren. Dazu wird eine In-vitro-transkribierte intronhaltige Modell-prä-mRNA mit Zellextrakten inkubiert, um die Anlagerung der snRNPs an die prä-mRNA zu erlauben. Unter geeigneten Bedingungen lassen sich so Spleißosomenkomplexe isolieren, die in einem bestimmten Funktionszustand arretiert sind. Damit war die Voraussetzung geschaffen, zunächst die Proteinzusammensetzung der wichtigsten Funktionsstadien humaner Spleißosomen mit massenspektrometrischen Mitteln zu charakterisieren. Diese Bestandsaufnahme ergab, dass während des gesamten Spleißzyklus insgesamt ca. 170 Proteine mit der in vitro assemblierten Spleißmaschinerie assoziiert sind, wobei jedes einzelne Funktionsstadium jeweils deutlich weniger Proteine enthält. Viele der Proteine weisen diverse post-translationale Modifikationen auf. Die massenspektrometrische Bestimmung der Phosphopeptide zeigte, dass ca. ein Drittel aller spleißosomalen Proteine phosphoryliert wird, wobei einige Phosphorylierungen nur in ganz bestimmten Funktionsstadien des Spleißosoms auftreten und als molekulare Schalter für Strukturänderungen des Spleißosoms fungieren. So phosphorylieren die SRPK2- und PRP4-Kinasen u. a. Proteine des U4/U6.U5-tri-snRNP-Partikels als Voraussetzung für seine stabile Integration ins Spleißosom.
Auch Spleißosomen der Fruchtfliege Drosophila melanogaster und der Bäckerhefe S. cerevisiae wurden isoliert und biochemisch charakterisiert. Dabei zeigen die Metazoen-Spleißosomen (Drosophila und humane) eine frappierende Übereinstimmung in ihrer Proteinzusammensetzung, sowohl was die Zahl als auch die Identität der Proteine betrifft. Die Gesamtzahl der Proteine, die im Hefespleißosom gefunden wurden, ist dagegen mit ca. 95 deutlich niedriger als die der Metazoen-Spleißosomen. Viele der Metazoen-spezifischen Proteine sind an der Regulation des alternativen Spleißens beteiligt, das in der Hefe kaum eine Rolle spielt. Für nahezu jedes spleißosomale Protein der Hefe ist ein evolutionär konserviertes Homolog in Metazoen-Spleißosomen vorhanden. Man kann daher davon ausgehen, dass wir es in der Hefe mit der evolutionär konservierten core-Spleißmaschinerie zu tun haben.

Vergleicht man die Proteinzusammensetzung der Funktionsstadien der Spleißosomen, ergibt sich ein erster Einblick in das dynamische Geschehen der spleißosomalen Proteinzusammensetzung (Abb. 3). So verlassen z. B. während des Übergangs vom prä-katalytischen in ein aktiviertes Spleißosom (s. Abb. 1) neben den snRNAs U1 und U4 ca. 35 Proteine das Hefe-Spleißosom, darunter alle U1 und U4/U6-snRNP- sowie einige U5-spezifische Proteine, d.h. die Strukturen einiger snRNPs ändern sich drastisch während der katalytischen Aktivierung des Spleißosoms. Andererseits werden mehr als 20 Proteine (darunter 12 neue) stabil ins aktivierte Spleißosom (Bact-Komplex) integriert. Mehreren dieser Proteine kommt eine besondere Bedeutung bei der gleichzeitig stattfindenden Umlagerung des RNA-RNA-Netzwerks des Spleißosoms zu (Abb 2). Aus den proteomischen Studien wird auch deutlich, dass die Dynamik in der biochemischen Zusammensetzung des Spleißosoms während seines Funktionszyklus ein von Hefe bis Mensch evolutionär konserviertes Designprinzip ist (Mitarbeiter: Anokhina, Bessonov, Dannenberg, Dönmez, Fabrizio, Hartmuth, Herold, Mathew, Schneider, Will, in Zusammenarbeit mit der AG H. Urlaub; MPI für biophysikalische Chemie).
In-vitro-Rekonstitution der katalytischen Phase von Hefe-Spleißosomen aus gereinigten Komponenten

Idealerweise würde man die Rolle einzelner Proteine oder snRNAs im Spleißmechanismus im Reagenzglas in einem vollständig aus den Einzelbausteinen rekonstituierten Spleißosom untersuchen. Die große Zahl der beteiligten Proteine und snRNPs und die Dynamik des Assemblierungsprozesses der Spleißosomen machen ein solches System, das ausschließlich rekombinant produzierte Proteine verwendet, aber nahezu unmöglich. Durch eine partielle Komplementationsstrategie im Hefesystem gelang es aber erstmals, die gesamte katalytische Phase mit gereinigten Komponenten in vitro zu rekonstituieren. Mit diesem System konnten die Einzelschritte der katalytischen Aktivierung des Spleißosoms analysiert und die Funktion einzelner Proteine während der katalytischen Phase aufgeklärt werden. Dieses führte auch zur Entdeckung und Funktionsbeschreibung eines neuen, für den ersten katalytischen Schritt notwendigen Proteins, Cwc25 (Abb. 1). Mit der Methode der Fluoreszenz-Korrelationsspektroskopie wurde die Dynamik der Proteine während der katalytischen Phase des Spleißosoms quantitativ und zeitaufgelöst erfasst. Weiterhin gelang es auch bereits, die Endphase des Spleißzyklus, d. h. die Dissoziation des gereinigten Intronlariat-Spleißosoms in seine Einzelteile, in vitro nachzuvollziehen (Mitarbeiter: Fabrizio, Fourmann, Odenwälder, Ohrt, Schmitzova, Warkocki, in Zusammenarbeit mit J. Enderlein, Universität Göttingen).
Struktur des katalytischen core RNPs von Spleißosomen
Zur Beantwortung der Frage, welche Proteine notwendig sind, um das katalytisch aktive RNA-Netzwerk des Spleißosoms zu generieren und aufrechtzuerhalten, wurden aufgereinigte humane C-Komplex-Spleißosomen stringenten biochemischen Bedingungen ausgesetzt, um so die Proteine zu identifizieren, die sehr fest ans Spleißosom gebunden bleiben. In 1-molarer Natriumchlorid-Lösung bleibt ein makromolekularer Spleißosomenkomplex einschließlich des RNA-Netzwerks intakt, der ca. 40 der insgesamt mehr als 100 Proteine des nativen C-Komplexes enthält. Durch Quervernetzungsstrategien konnten die Proteine identifiziert werden, die in direktem Kontakt mit katalytisch wichtigen RNA-Elementen stehen und damit im Herzen des katalytischen Zentrums liegen. Chemische Modifikationsstudien der RNA sowie RNA-RNA Quervernetzungen in humanen Spleißosomen lieferten ein detailliertes Bild über das RNA-RNA-Interaktionsnetzwerk im katalytisch aktiven Spleißosom. Auf der Basis dieser Strukturdaten konnte ein erstes 3D-Modell des RNA-RNA-Netzwerks im katalytischen Zentrum erstellt werden (Mitarbeiter: Anokhina, Bessonov, Fabrizio, Hartmuth, Rasche, Will, in Zusammenarbeit mit der AG H. Urlaub (Max-Planck-Institut für biophysikalische Chemie) und E. Westhof (IBMC Strasbourg).
Untersuchungen zur 3D-Struktur isolierter snRNPs und Spleißosomen

Ein tiefes mechanistisches Verständnis der Funktionsweise des Spleißosoms und seiner strukturellen Dynamik ist ohne ein Bild der Raumstruktur der wichtigsten Funktionsstadien des Spleißosoms nicht denkbar. Die Kryo-Elektronenmikroskopie ist die Methode der Wahl, um erste Einblicke in die Architektur hochmolekularer und dynamischer Maschinen wie der Spleißosomen zu bekommen. Als Voraussetzung hierfür wurden strukturstabilisierende Methoden (GraFix genannt) entwickelt, welche die Integrität von Spleißosomen während der Präparatherstellung gewährleisten. Bisher wurden die 3D-Strukturen der wichtigsten Funktionsstadien des humanen Spleißosoms bei Auflösungen von ca. 20-30 Å rekonstruiert. Mittels spezifischer Marker, wie Antikörper oder RNA-Sonden, wurden zudem Informationen erhalten, welche die Position einzelner Proteine im Spleißosom genauer beschreiben. Auch über die Bestimmung der 3D-Struktur isolierter snRNPs und von Subkomplexen des Spleißosoms und deren virtuelles Einpassen in die native Spleißosomenstruktur wurden wichtige Informationen zur Spleißosomen-Architektur erhalten. So konnte die Lage bestimmter Proteingruppen und des katalytischen Zentrums im nativen C-Komplex-Spleißosom bestimmt werden (Abb. 4A).
Ein längerfristiges Ziel liegt darin, die Arbeitsweise der Spleißmaschinerie auf atomarer Ebene zu verstehen. Über rekombinant hergestellte Proteine konnten bereits mehrere Proteinkomplexe des Spleißosoms durch Röntgenkristallografie atomar aufgeklärt werden, u. a. auch der für die Spleißosomen-Aktivierung wichtige Komplex aus U4-RNA und den Proteinen hSnu13 und hPrp31 (Abb. 4B). Komplementär zu dem rekombinanten Weg werden auch aus humanen Zellen isolierte native snRNPs der Röntgenkristallstruktur-Analyse zugeführt. So ließ sich die Kristallstruktur des humanen U1-snRNP-Partikels bei 4,2 Angström Auflösung aufklären (Abb. 4C). Die U1-snRNP-Struktur zeigt u. a. die große Vielfalt verschiedenartiger Wechselwirkungen zwischen dem Ring der sieben Sm-Proteine und der snRNA in einem nativen Partikel, einer Grundstruktur, die auch den snRNPs U2, U4 und U5 gemeinsam ist. Ein Schwerpunkt der Arbeiten am MPI für biophysikalische Chemie liegt in den nächsten Jahren darin, isolierte Spleißosomen in definierten Funktions-Zuständen zu kristallisieren, wofür alle methodischen und präparativen Voraussetzungen inzwischen geschaffen wurden (Mitarbeiter: Fabrizio, Häcker, Kastner, Wolf, in Zusammenarbeit mit der AG H. Stark (MPIbpc) und M. Wahl (MPIbpc/FU Berlin).